參賽團隊名稱:拉拉的奇幻世界
提案動機
台灣在1965年時本土瘧疾已經根除,但衛生福利部疾病管制署副署長莊人祥說,在國際上瘧疾仍然是一個相當重要的疾病問題。台灣現在一年還是有10~40例境外瘧疾病例,原因許多是在從非洲經商回台的人士身上檢出。透過文獻蒐集與Kaggle資料集發現,有許多瘧疾血液樣本資料集,可採用機器學習來進行瘧疾血液的診斷進行分類預測。
本專案希望能透過較多種類的瘧疾血液資料集建構一個,較為廣泛使用情境的瘧疾血球辨識系統,最後透過OpenVINO的邊緣運算架構,在沒有網路的環境下幫助進行瘧疾診斷辨識的工作。
使用之軟硬體:
A. 硬體
-
-
-
- 環境測試:
- CPU: 11th Gen Intel(R) Core(TM) i5-1145GRE @ 2.60GHz
- RAM: 16G
- 運作環境:
- 環境測試:
-
-
-
-
-
-
- Raspberry PI4 – 4GB
- Storage:32GB
- VPU:Intel® Movidius™ Myriad™
-
-
-
B. 軟體
-
-
-
- PC:
- Ubuntu 20.04.3 LTS
- Docker
- OpenVINO 21.4.582
- Raspberry PI:
- Raspbian* Buster, 32-bit
- 模型訓練使用工具平台:
- Google Colab
- TensorFlow 2.7
- Keras
- Python 3.8.10
- PC:
-
-
解決方案設計構想-開發目標:
-
- 運用技術:機器學習 + 電腦視覺
- 技術定位:透過邊緣運算進行機器學習分類,判斷血液樣本是否感染瘧原蟲
- 邊緣運算:採用OpenVINO運作架構,於無網路狀態下執行血球辨識
- 機器學習:將血液樣本透過Tensorflow或Yolov4訓練之機器學習模型,
轉換為OpenVINO可使用之Inference Reference(IR)模型,於OpenVINO下執行運作。
資料來源:
-
- Malaria主要分為P.falciparum惡性瘧、P.vivax間日瘧、P.malariae三日瘧、P.ovale卵形瘧。
- 淺血樣本(thin smears)à共229影像,包含Plasmodium falciparum、P. malariae、P. ovale、P. vivax
- 厚血樣本(thick smears)à1182 影像: Plasmodium falciparum
- 淺血樣本(thin smears)à 27,558 個血球標注(已感染與未感染紅血球資料): Plasmodium falciparum
- 淺血樣本(thin smears)à655個影像(17類): Plasmodium falciparum、P. malariae、P. ovale、P. vivax
檢體資料來源可能來自多種不同的過程:
實際世界的檢體來源過程充滿多樣性,各種文獻討論的研究方法也不同,如果能將資料標注的成果進一步整合,將不同瘧原蟲物種以及淺血/厚血的資料訓練成更完整的模型,將能更大幅度的擴展瘧疾檢體辨識的應用性。
解決方案設計構想-機器學習訓練模型:
-
- 文獻顯示2018年美國國家衛生研究院提供瘧疾血球樣本,透過ResNet-50的辨識學習結果相對有比較好的預測率。
- 針對其他種類的虐原蟲資料,將參考同樣方式在進行較多種類虐原蟲資料集訓練時,是否能持續維持預測結果的效能。
解決方案設計構想-將訓練結果轉換為Inference Reference(IR)模型:
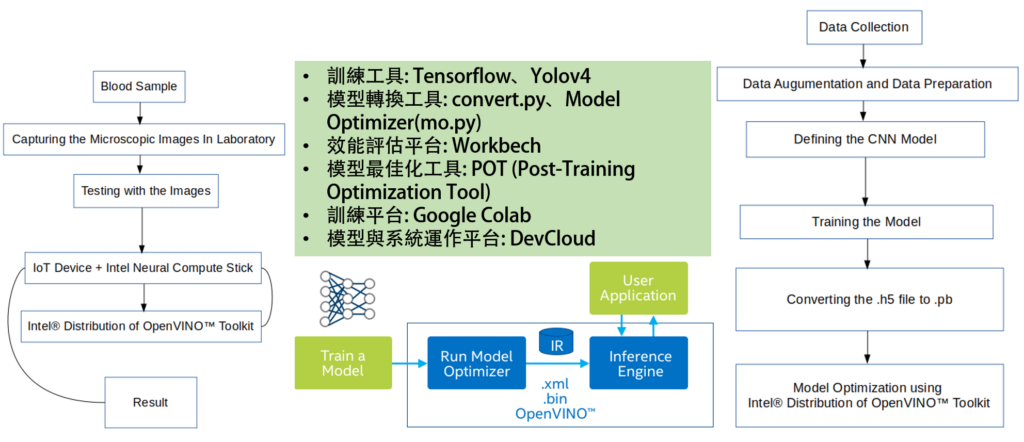
- 本次專案計畫透過資料機器學習訓練、將訓練成果轉換為IR模型結構、最後透過OpenVINO執行,將使用Python作為系統開發語言,將IR檔載入OpenVINO執行
-
- 訓練工具: Tensorflow、Yolov4
- 模型轉換工具: convert.py、Model Optimizer(mo.py)
- 效能評估平台: Workbech
- 模型最佳化工具: POT (Post-Training Optimization Tool)
- 訓練平台: Google Colab
- 模型與系統運作平台: DevCloud
 解決方案設計構想-將模型成果配置於Raspberry PI + NCS2運行:
解決方案設計構想-將模型成果配置於Raspberry PI + NCS2運行:
-
- Raspberry PI + NCS2運作OpenVINO將可有效的達成設備的微型化、輕量化、成本降低、可快速擴展預測推論硬體數量等優點(搭配USB HUB使用多個VPU)。
 商業價值與可行性:
商業價值與可行性:
-
- 由Malaria Hero的需求與經驗中,可以清楚知道瘧疾輔助判斷系統對於大規模擴散疫情的判斷過程中,產生很大的作用。
- 將預測模型轉換至OpenVINO可使用之IR模型,最後將邊緣運算版本安裝至Raspberry PI與NCS2的裝置,可以將此裝置開發為隨身攜帶的瘧疾檢測隨身版本。
- 一方面大幅度降低設備的成本,同時可攜性也提高非常多,對於日後擴展為血液寄生蟲篩檢的行動裝置平台,也提供了快速轉移的基礎架構。




