參賽團隊名稱:嘉縣一中夢之隊
由於獲得大量帶有標註資料的醫學影像並不容易,因此,本專案採用了自監督式學習方法架構,從少量醫學資料中學習醫學影像的良性息肉與惡性息肉的特徵表示方法,可有效部屬Edged AI裝置,結合OpenVINO Toolkit進行息肉分類。
作品簡述
- 本專案成果系統設計概念如下圖1,Pretext Task部分,輸入使用大量未標註之醫學影像讓複雜的模型(Teacher Model)學習醫學影像特徵表示(feature representation)以利進行分類學習,在這邊我們分類有兩種類別,一種是良性增生性息肉(HP),另一種是惡性腺瘤性息肉(TA),在分類任務前,我們會使用對比式學習來做到損失函數來反向傳播最佳化模型編碼器(ConvNet-encoder)。
- 如圖2為對比式學習架構圖,這個對比式學習架構圖我們會將原始影像X,透過將相似的特徵元素X1與X2聚類,並且同時將不同的特徵元素分離,學習其中不同的特徵描述方式Z1與Z2,這樣的方法可做到有效虛擬標籤(pseudo label)與模型優化的結果。
- 而Downstream部分,透過Knowledge Transfer傳到Student Model,這個過程就像老師再教學生知識,將模型壓縮為輕量化的過程,然後再將少量有標註資料輸入該輕量化模型進行測試,達到我們正確的分類結果。實驗部分我們測試了四個模型,分別為ResNet-18、ResNet50V2、VGG16、MobileNetV3small,我們使用的公開資料集為KvasirV2[1],此數據集我們使用如下類別(class):
-
- normal:正常腸道。
- polyps:息肉,腸內隨著黏膜長出而可檢測到的病變。
- dyed-lifted-polyps:染色息肉,通過注射生理鹽水和靛藍胭脂紅使其變明顯的息肉。
- 實驗以上所述資料集與不同模型訓練,其中以downstream 部分ResNet-18效果最好,結合OpenVINO Toolkit,最後評比指標可達到近乎100%的分類結果。
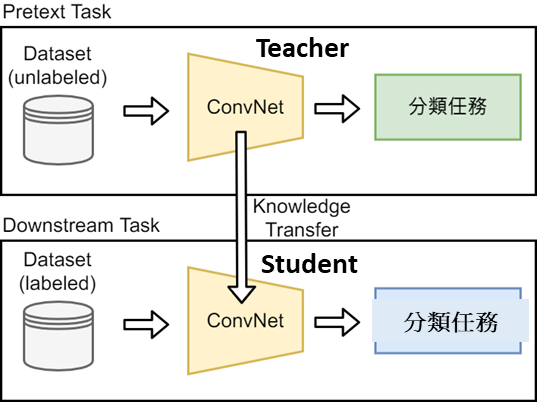
圖1. 知識蒸餾系統設計概念圖:上方Pretext Task部分使用unlabeled data醫學影像資料集透過Teacher Model學習特徵表示,下方Downstream Task部分使用labeled data影像透過knowledge Transfer 形成輕量化模型(Student Model)進行分類任務。
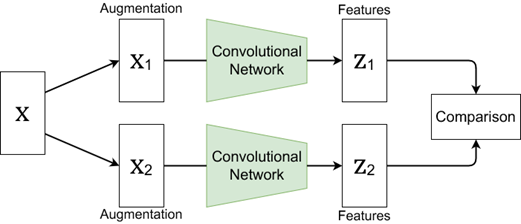
圖2. 資料擴增結構圖(Data Augmentation Structure):X為原始影像,透過將相似的特徵元素X1與X2聚類,同時將不同的特徵描述分離,學習其中不同的特徵描述(feature representation)方式Z1與Z2進行對比學習(contrastive learning)
提案動機
- 大腸息肉是因為細胞增生的突起組織,大部分息肉小於一公分,通常為良性組織,但少數息肉發現時,已經變大發生惡性變化,也就是大腸癌。如果在息肉尚未癌變或早期癌變時就切除,則大腸癌的發生率便可降低。
- 大腸鏡檢查易受到操作者的經驗影響,與器具的照光不足、過度曝光(白光)、移動跟液體所造成的影像模糊,加上醫師的視覺疲勞等因素,將直接影響診斷的準確性,我們可將有效分類,對醫生是莫大的幫助。
- 而近期深度學習方法在應用上越來越成熟,且圖像辨識的辨識率在特定資料及下,會有極佳的表現,我們將採用圖像分類模型配合知識蒸餾技術(Knowledge Distillation; KD)將模型輕量化來輔助醫師的息肉影像分類與判斷,可提高惡性息肉的檢出率,減少漏診與提高診斷效率。
- 輕量化模型(壓縮模型)代表可部署在裝置更小的設備上,若醫師有一台輕量化可偵測與分類良性與惡性息肉的小設備,例如:手機裝置,就宛如身邊有一個小助理可協助判斷。
解決方案
- 使用的資料集KvasirV2[1]:
- normal:正常腸道。
- polyps:息肉,腸內隨著黏膜長出而可檢測到的病變。
- dyed-lifted-polyps:染色息肉,通過注射生理鹽水和靛藍胭脂紅使其變明顯的息肉
- 知識蒸餾的優點知識蒸餾由2015年Hinton的論文「Distilling the Knowledge in a Neural Network」推廣,他們採用teacher/student師徒概念的框架來實現,由teacher模型先訓練好權重後,再抽取(蒸餾)精華作為student模型的訓練教材,讓student也能達到比美teacher的效果,如圖3、圖4所示。
要讓邊縁設備擁有高的辨識率,一種是使用API來提供遠端服務存取是方式之一,但是這樣會讓邊縁設備增加很多限制:網路功能速度的延遲…等等,因此,模型縮小化deploy到邊縁裝置上,變成另外一種解決方案,最常使用的方便法,便是Knowledge distillation與Model compression,不過,雖然Knowledge distillation比起後者較為簡單易行,但限制也較多(侷限於分類任務的模型)。
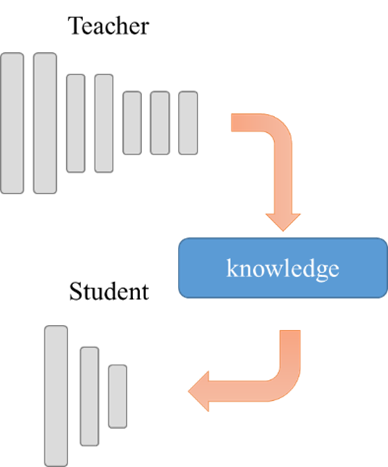 圖3. teacher/student師徒概念的模型框架來實現知識蒸餾結果
圖3. teacher/student師徒概念的模型框架來實現知識蒸餾結果
圖4. student模型的訓練教材,讓student也能達到比美teacher的效果
- 系統技術架構:如下圖5細節架構說明,在輔助任務(Pretext Task)我們使用大量未標註的醫學息肉影像資料來進行特徵學習,而ConvNet(Resnet-50)輸出的特徵圖(息肉特徵)會輸入至Prediction Head進行比對式學習(Contrastive learning)來調整網路神經參數,調整完後的ConvNet(Resnet-50)會進行知識蒸餾的方式下傳至Downstream的ConvNet(Resnet18),下游( Downstream)我們實驗使用不同模型的神經網路,而其中Resnet18得到的效果最好,證明我們使用知識蒸餾方式有效學習到息肉特徵。Downstream Task 我們同時使用少量標註的資料當成是我們的輸入資料,可正確進行良性與惡性息肉的分類。兩階段進行學習效果宛如Pretext Task 是一個老師,Downstream task 是一個學生再學習老師知識的傳遞,學生角色我們使用OPENVINO來進行推論與優化輸出量化(quantization)至IR檔至推論引擎(Inference Engine)來判斷息肉是否為良性還是惡性。
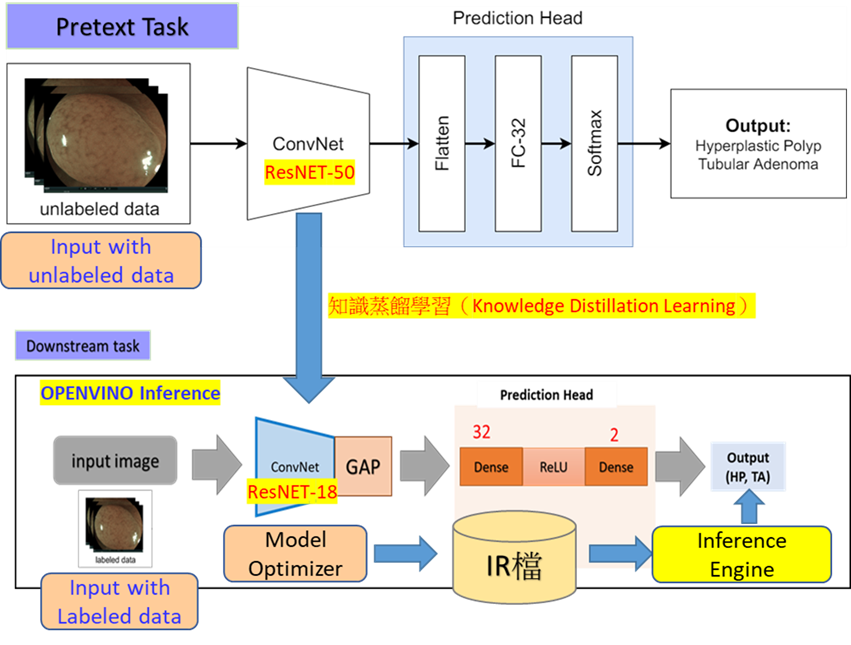
圖5. 知識蒸餾學習結合自監督式學習息肉分類架構圖
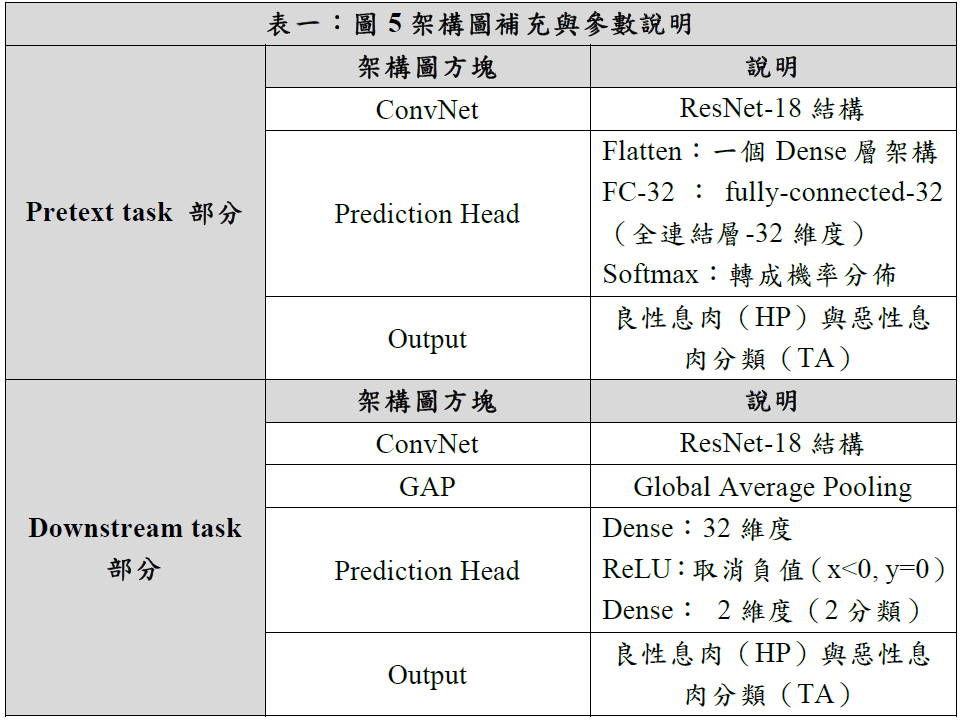
※下表一為圖5架構圖介紹:
※ 補充Pretext task部分:對比學習(contrastive learning)與資料擴增(data augmentation)架構細節說明:
如下圖6是對比學習示意圖,我們會將原始影像(息肉)X進行資料擴增,例如:上下左右翻轉。擴增成兩張圖片X1與X2輸入Convolutional Network得到Z1與Z2特徵向量表示,因相同類別其特徵向量分佈會是相似的,因此可以用這樣的觀點去判別相同類別還是不同類別,我們將此觀點套用至我們良性與惡性息肉分類。
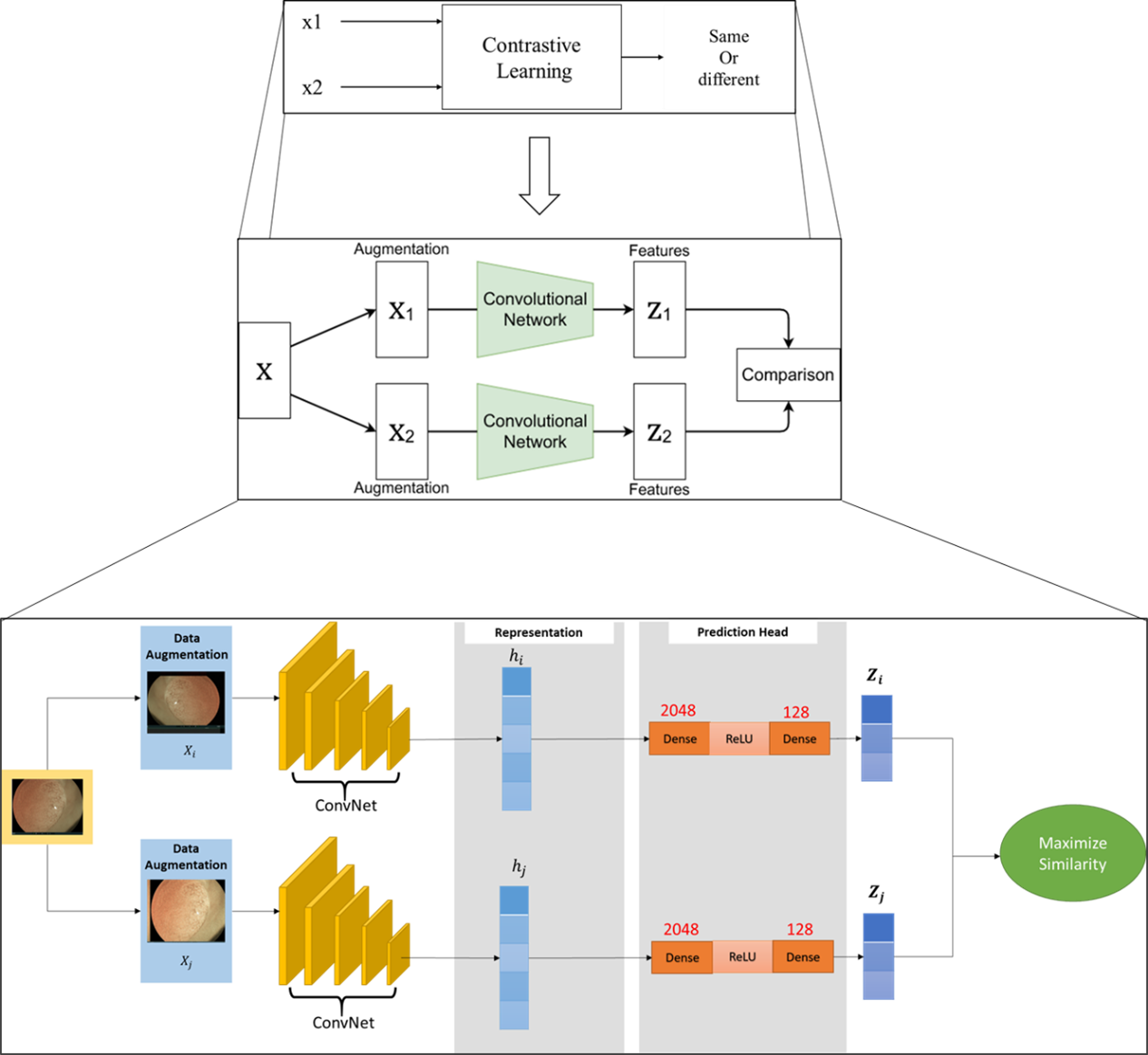
圖6. 息肉對比式學習(contrastive learning)結合資料擴增細部架構圖
- Knowledge Distillation的作法知識蒸餾其實觀念和作法很基本,什麼是知識蒸餾呢?我們可以想成:老師所傳授給學生的「知識」其實只是訓練後各類別的預測機率,透過「蒸餾」讓這個知識更淺顯易懂的傳授給學生。 以我們息肉分類來說,有2種分類,在訓練模型時我們會給予如下表二目標:
表二

但是,若我們能給予如下表三的機率分佈,而非單純的對與不對:
表三

這樣的數字其實提供了更多的隱含資訊(Dark knowledge),例如我們可以查知各類別彼此相異/相似的程度,在訓練過程中增加更多的判斷條件。但是,最大問題在於,一般分類模型所輸出 的預測是使用softmax函數,因此老師所預測出的結果其各類別間的概率分佈是具有很高極端值的機率,也就是正確類別的機率相當接近於1,而所有其它類別的機率都非常接近0,因此Hinton團隊(2015年)引入了「softmax temperature」的概念,也就是,透過一個數值T把這些輸出的機率平坦化soften these probabilities。我們知道Softmax是將多個類別機率輸出映射到(0,1)區間內,如下公式(1)與(2):
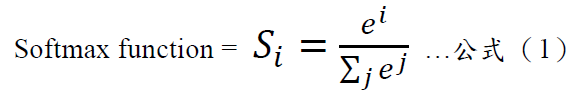
參數說明:其中𝑖表示第𝑖個類別,𝑗表示所有類別加總,𝑒表示指數函數。
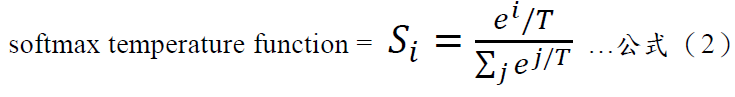
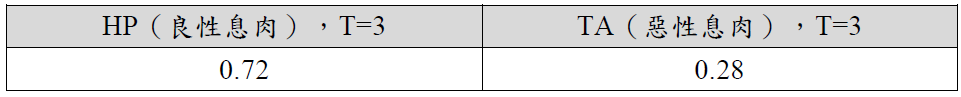
如果T為1,就等於與原本的Softmax公式相同沒有變化,一般來說,T值會設到3以上,那麼就可以將上述的類別機率變成如下,等於是隱含的訊息顯露更多出來,我們稱為Softened probabilities或Dark knowledge,而使用這個資訊來訓練模型的方法,則稱為distillation蒸餾,如下表四T=3所得出來的值。
表四
5. OPENVINO套件使用架構
由上述訓練完成的模型輸出ONNX後,再投入MODEL OPTIMIZER,輸出成IR模型,最後投入推論引擎 Inference Engine產生最終的分類輸出,如圖7,MODEL OPTIMIZER輸出 IR,如圖8。 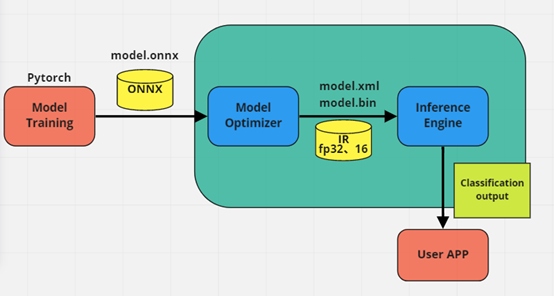
圖7. 輸出ONNX後,再投入MODEL OPTIMIZER,輸出成IR模型
.jpg)
圖8. MODEL OPTIMIZER輸出IR檔
軟硬體規格
硬體:

EC70A-TGU
軟體:
- python3.9
- pytorch 1.10
- Intel Openvino Toolkit
成果介紹短片:
在訓練期間我們採用ResNet-18作為核心模型,能夠提供參數量更小與運算速度更快的特性,並且加入Knowledge Distillation(KD)來輕量化模型重點物件捕抓的能力。透過我們模型架構,可以有效壓縮模型並將大腸病變分類出來,接著再利用GRAD-CAM可解釋化模型說明重要特徵,以利醫生能夠快速確認問題。透過OPENVINO壓縮降精準度推論方法,我們模型不會失去精準度,因為已經蒸餾輕量化,可以非常快速的將分類結果運算出來,包裝成DOCKER後可以輕鬆部屬到任何OPENVINO支援的edged裝置上。
成果介紹
本成果使用Recall, Precision, F1-socre評估模型的效能。其中研究中共有兩個類別,分別為Hyperplastic Polyp(在此視為1, positive)及Tubular Adenoma(在此視為0, negative),如表五。
表五. 混淆矩陣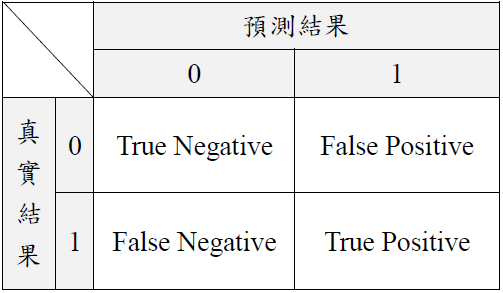
混淆矩陣內各矩陣定義如下:
- True Positive(真陽性,TP):實際值為陽性,模型預測仍為陽性(預測正確)。
- True Negative(真陰性,TN):實際值為陰性,模型預測仍為陰性(預測正確)。
- False Positive(偽陽性,FP):實際值為陰性,模型預測仍為陽性(預測錯誤)。
- False Negative(偽陰性,FN):實際值為陽性,模型預測仍為陰性(預測錯誤)。
以上混淆矩陣跟延伸如下公式:
- Recall是所有實際上為此類別中,模型正確預測的比例,公式(3):

- Precision是所有類別預測中,預測為陽性的樣本中,實際為陽性的比例,公式(4)如下:

- F1-score為能同時考慮Precision及Recall兩種指標,平衡的反映類別預測的精確度,公式(5)如下:
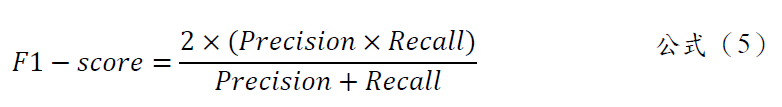
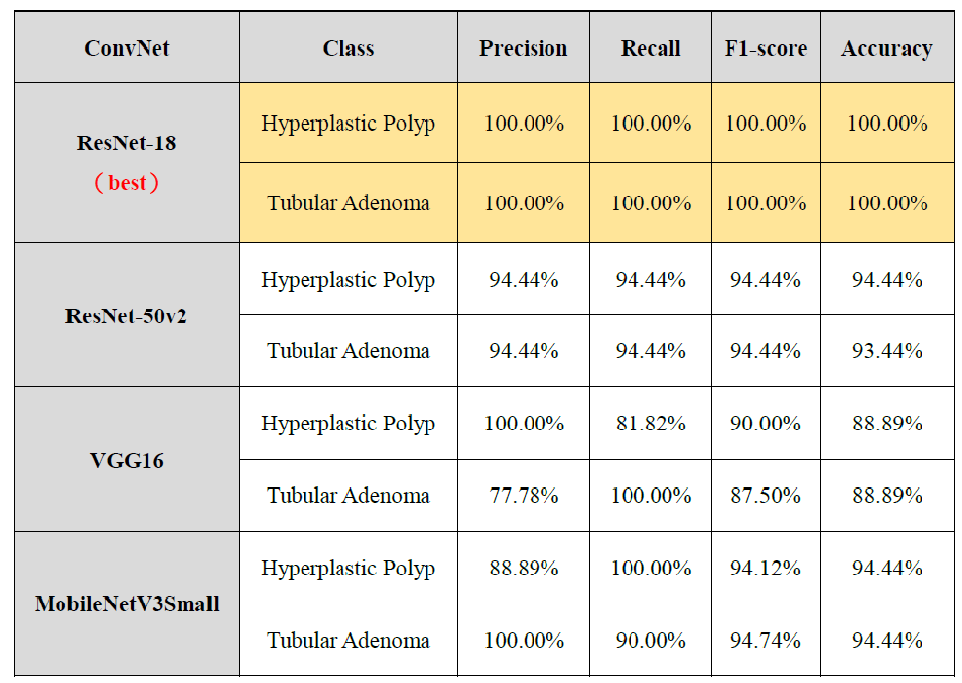
1.分類結果評估指標(以ResNET-18知識蒸餾效果最佳),如下表六:
表六:分類結果評估指標(ResNet-18為最好結果)
2.分類結果展示圖(以ResNET-18知識蒸餾效果最佳),如下圖9:
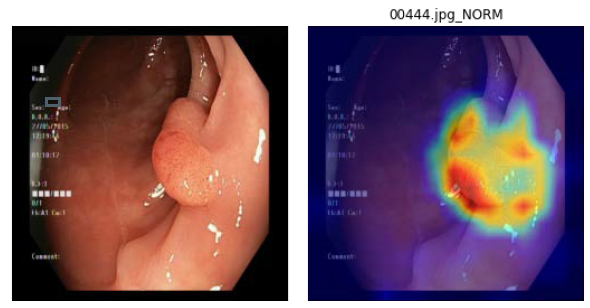
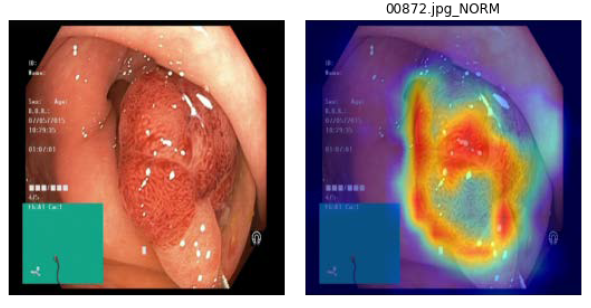
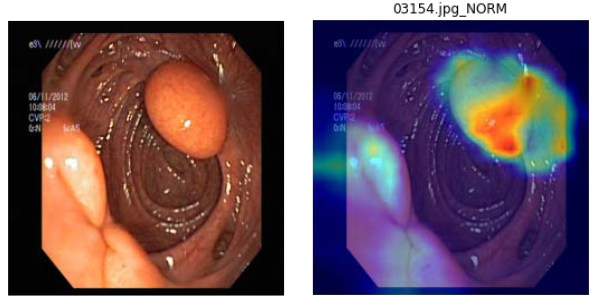
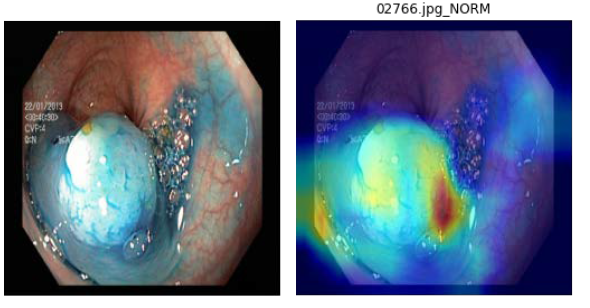
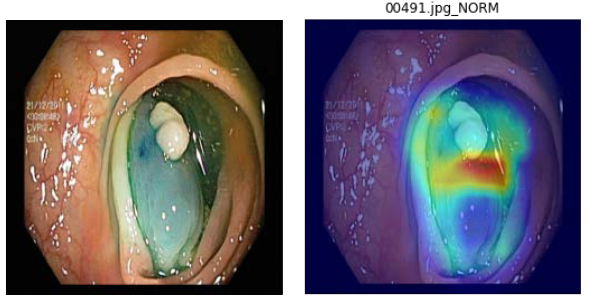
圖9. 左為原圖,右為蒸餾學習的結果圖(student學習結果)
創新價值與商轉可行性
- 客群:
- 實習醫生
- 醫生
- 大型醫院
- 生醫公司(筑波科技)
- 創新價值:
- 醫師疲憊情況下:在醫療環境長期工時下,醫師可能處於疲勞狀況下,則可結合本知識蒸餾演算法,協助自動息肉分類,並分析是否為惡性息肉。
- 實習醫生(經驗不足):剛畢業的醫師需要至醫院駐診實習,在經驗不足的情況下,可使用本視覺圖像偵測演算法協助判斷。
- 本演算法可有效壓縮模型卻不失精準度之息肉分類結果,未來將結合關注度(attention)機制,擬合更正確的息肉位置,可將技轉給生醫公司,例:筑波生醫公司、國外的生醫公司。
- 可行性:
- 醫生可減少看診時間
- 快速判斷良/惡性息肉類別
- 提昇診斷效率
- 協助實習醫生辨別息肉
團隊自介

附件與參考文獻:
- kvasir-v2 Dataset-https://www.kaggle.com/datasets/plhalvorsen/kvasir-v2-a-gastrointestinal-tract-dataset
- Colorectal Polyp Detection in Real-world Scenario: Design and Experiment Study
- Real-time detection of colon polyps during colonoscopy using deep learning: systematic validation with four independent datasets
- D Geoffrey Hinton, Oriol Vinyals, Jeff Dean “Distilling the Knowledge in a Neural Network” https://doi.org/10.48550/arXiv.1503.02531
- G. E. Hinton, T. J. Sejnowski, T. A. Poggio, eds. Unsupervised Learning: Foundations of Neural Computation, MIT Press, 1999.
- T. Ojala, M. Pietikäinen, and D. Harwood, “A Comparative Study of Texture Measures with Classification Based on Feature Distributions,ˮ Pattern Recognition, vol. 29, no. 1, pp. 51-59. Jan. 1996.
- N. Dalal and B. Triggs, “Histograms of oriented gradients for human detection,” In Proc. Comput. Vis. Pattern Recognit, (CVPR), San Diego, CA, USA, 2005, pp. 886–893.
- Krizhevsky, I. Sutskever, and G. E. Hinton, “ImageNet classification with deep convolutional neural networks,” In Proc. Neural Information Processing Systems , (NIPS) , 2012, pp.1090–1098.
- K. He, X. Zhang, S. Ren, and J. Sun, “Deep residual learning for image recognition,” In Proc. Comput. Vis. Pattern Recognit, (CVPR), Las Vegas, NV, USA, 2016, pp. 770-778.
- S. J. Pan and Q. Yang, “A survey on transfer learning,” IEEE Trans. Knowledge and Data Engineering, Vol. 22, No. 10, pp. 1345-1359, Oct. 2010.
- Ribeiro, A. Uhl, and M. Häfner, “Colonic polyp classification with convolutional neural networks,” In Proc. International Symposium on Computer-Based Medical Systems (CBMS), Dublin, Ireland, Belfast, Northern Ireland, 2016, pp. 253–258.



Great job!
Nice
Good👍
Masterpiece
awesome!
太強了~~好棒
Great
印象中上一屆實作組已經做過類似的課題…
就只是換了個模型訓練的方式而已。
1. 醫學辨識上更注重的應當是準確度,這方法顯然是以精準度作為犧牲點的架構…
2. 以數據和描述來看,似乎有矛盾?資料集描述最少有3個類別(看影片裡面的輸出甚至到達了9個,但貴組顯示的只有兩個?)
3. 這分類雖號稱有100%的結果,但是有瑕疵的。結果顯得的類別均為有息肉的類別,只是名詞不同。以貴組顯示的binary結果,那遇到沒有息肉的輸入,顯然就有落差了。這點上一屆大腸息肉的分析做得比較完整,可以參考。
4. 在你們推出的架構上,只有簡短的敘述了概念,並沒有提及更深層的解析,如 feature maps 矩陣的轉換,顯然還有待加強。然而,這也沒有導出 openvino的優勢,看上去更像是為了比賽才讓openvino做最後的配合…