參賽團隊名稱:嫌dis老孫
作品簡述:
- 本研究內容提出自監督式學習架構(self-supervised structure),結合正相關圖片(positive image)增強物件偵測的關注度,而本物件目標即為大腸癌的前身「息肉(Polyp)」,與OpenVINO技術整合,可正確協助醫生惡性息肉的檢出率,減少漏診,提高診斷效率。
代表圖片(如下圖1描述):
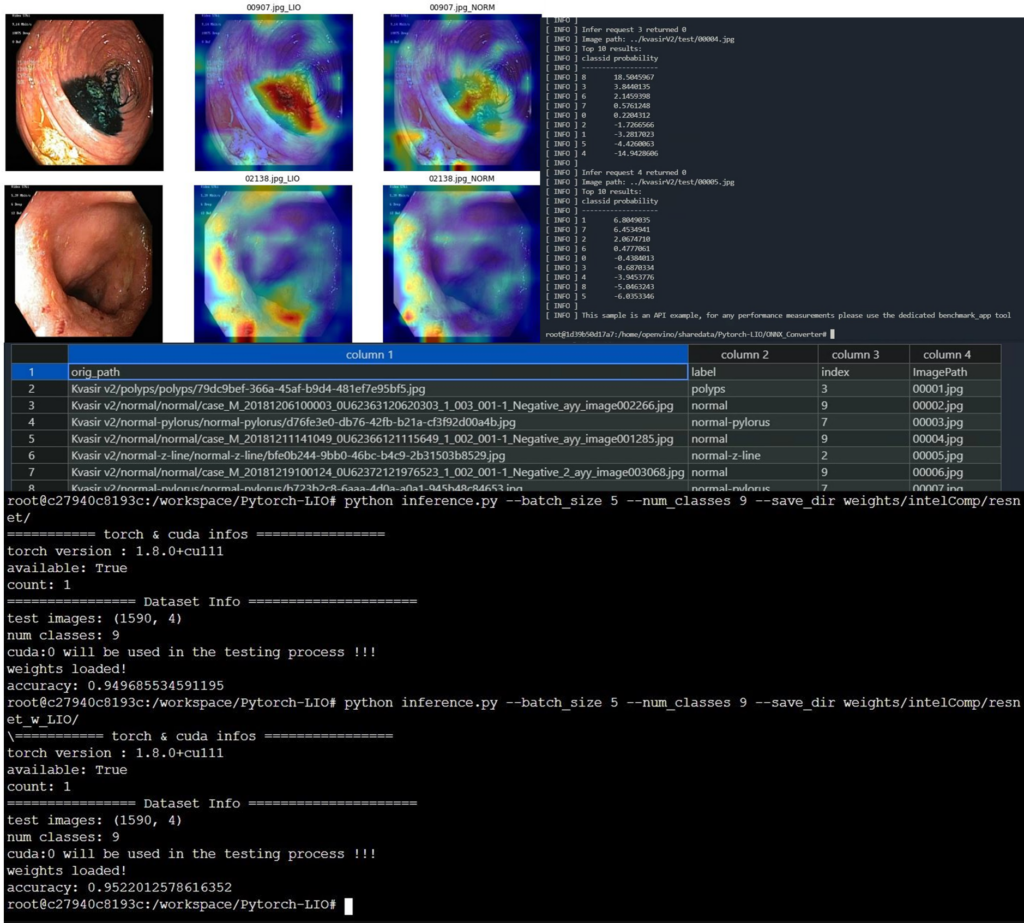
圖1. 左上角三張比較圖,第一張為原始圖片,中間為本研究內容提出自監督式架構關注度(self-supervised structure attention)方法,右圖為傳統CNN物件偵測方法。
提案動機:
- 在台灣,直腸癌是國人死因的第一位,如何有效地且早期地識別大腸息肉是非常重要的(如下影片:2021/12/24 TVBS新聞報導)。大腸息肉是因為細胞增生的突起組織,大部分息肉小於一公分,通常為良性組織,但少數息肉發現時,已經變大發生惡性變化,也就是大腸癌。如果在息肉尚未癌變或早期癌變時就切除,則大腸癌的發生率便可降低。
- 大腸鏡檢查易受到操作者的經驗影響,與器具的照光不足、過度曝光、移動跟液體所造成的影像模糊,加上醫師的視覺疲勞等因素,將直接影響診斷的準確性。
- 因此本動機將提出自監督式學習架構(self-supervised structure),結合正相關圖片(positive image)增強物件偵測的關注度,而本物件目標即為大腸癌的前身「息肉(Polyp)」來正確協助醫生惡性息肉的檢出率,減少漏診,提高診斷效率,呈現方式使用Grad-cam套件熱圖分佈來明顯息肉的位置。
影片連結(TVBS新聞網提供,2021/12/24報導)
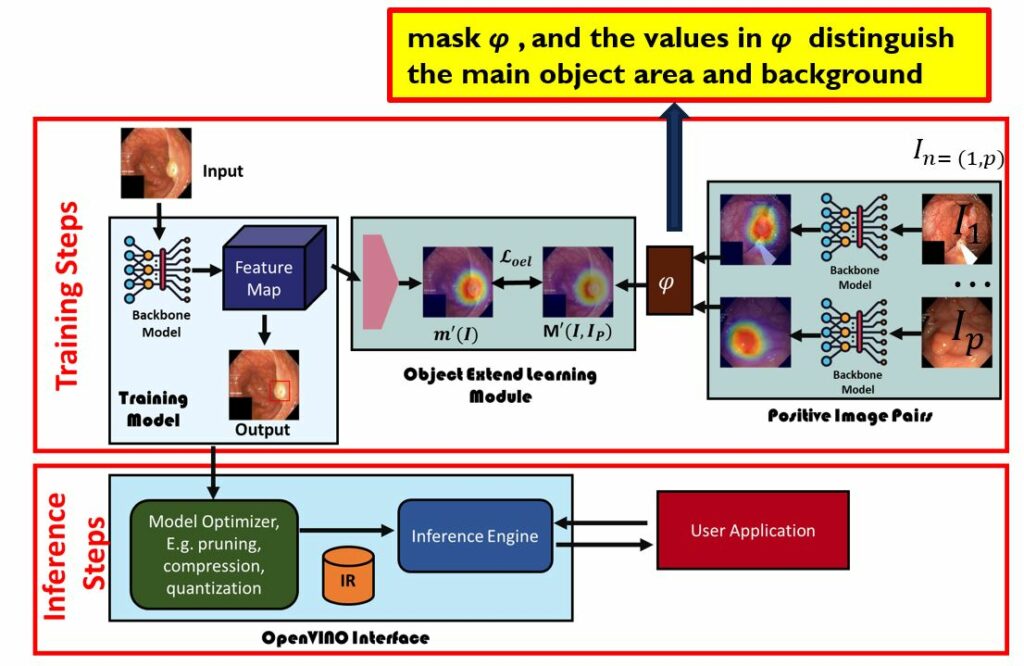
解決方案(如圖2技術架構說明):
圖2. 技術架構說明
在開發過程中所用軟硬體如下:
硬體:
- Lex-System SKY 3 – 3I110HW
- 11th Gen Tiger Lake-UP3 i7 /i5 /i3, Celeron CPU
- 2 x DDR4 SODIMM socket, Max. 64GB,
- Multiple Independent display: VGA & HDMI
- 3 x Intel GbE LAN, 4 x USB 2.0, 4x COM, Audio
- 1 x Mini PCIe, 1 x SIM , 2 x M.2, DI/O
- 硬體產品介紹:
- 影片連結:
軟體:
- python3.8
- pytorch 1.8
- Intel Openvino Toolkit
創新價值與商轉可行性:
| 客群 | 貢獻價值 | 可行性 |
|
|
|
成果:
成果介紹短片:
成果介紹:
測試過程(Inference Process)中,我們會將backbone model輸入到OpenVINO介面上,透過ONNX轉檔,這也使得許多沒必要的參數可以被省略掉,降低晶片需要存放模型的空間。OpenVINO會對訓練好的backbone model進行model optimization,如quantization在存入IR檔,放入Inference Engine,我們只要將息肉(Polyp)圖片(kvasir-v2 Dataset – 參考資料2)輸入,就可以做到realtime inference,供使用者(醫生)使用。
參考資料:
- The Kvasir-SEG Dataset
- kvasir-v2 Dataset
- Look-into-Object: Self-supervised Structure Modeling for Object Recognition
- Colorectal Polyp Detection in Real-world Scenario: Design and Experiment Study
- Real-time detection of colon polyps during colonoscopy using deep learning: systematic validation with four independent datasets
- G. E. Hinton, T. J. Sejnowski, T. A. Poggio, eds. Unsupervised Learning: Foundations of Neural Computation, MIT Press, 1999.
- A. Gosavi, Simulation-based Optimization: Parametric Optimization Techniques and Reinforcement, Springer, 2003.
- T. Ojala, M. Pietikäinen, and D. Harwood, “A Comparative Study of Texture Measures with Classification Based on Feature Distributions,ˮ Pattern Recognition, vol. 29, no. 1, pp. 51-59. Jan. 1996.
- N. Dalal and B. Triggs, “Histograms of oriented gradients for human detection,” In Proc. Comput. Vis. Pattern Recognit, (CVPR), San Diego, CA, USA, 2005, pp. 886–893.
- A. Krizhevsky, I. Sutskever, and G. E. Hinton, “ImageNet classification with deep convolutional neural networks,” In Proc. Neural Information Processing Systems , (NIPS) , 2012, pp.1090–1098.
- K. He, X. Zhang, S. Ren, and J. Sun, “Deep residual learning for image recognition,” In Proc. Comput. Vis. Pattern Recognit, (CVPR), Las Vegas, NV, USA, 2016, pp. 770-778.
- S. J. Pan and Q. Yang, “A survey on transfer learning,” IEEE Trans. Knowledge and Data Engineering, Vol. 22, No. 10, pp. 1345-1359, Oct. 2010.
- M. Everingham, S. M. A. Eslami, L. V. Gool, C. K. Williams, J. Winn, and A. Zisserman, The PASCAL voc2012 challenge results.
- A. Neubeck, L. V Gool, “Efficient non-maximum suppression,” In Proc. International Conference on Pattern Recognition (ICPR), HK, China, 2006, pp. 850-855.
- E. Ribeiro, A. Uhl, and M. Häfner, “Colonic polyp classification with convolutional neural networks,” In Proc. International Symposium on Computer-Based Medical Systems (CBMS), Dublin, Ireland, Belfast, Northern Ireland, 2016, pp. 253–258.




太多棒了
很棒且實際的作品
很實用的作品
很棒的作品!濟世救人!
怎麼那麼強
實用
很棒的題目,值得深入研究產品化
厲害的作品
👍🏻👍🏻👍🏻
強👍🏻👍🏻
👍
專業 好棒好棒!
很厲害唷! 棒棒棒!
厲害!
台灣之光
超棒的作品
棒
太強了
專業高手👍
好讚的應用 台灣有你真好